RNA-Seq(RNAシーケンス/トランスクリプトーム解析)受託サービス
特徴

RNA-Seq(RNAシーケンス)受託サービスの特徴
- 専門のスタッフがイルミナ社シーケンサーを用いてSOPに準じて国内ラボで実験解析を行います。
- 衛生検査所の資格を有し、製薬企業をはじめ大学等の研究機関からのご依頼も多数あり、臨床研究の実績もあります。
- 1ng以下の微量サンプル、FFPE検体など、サンプルの性状に沿って実験計画から解析までサポートします。一度ご相談ください。
サンプルの量と品質が十分に基準を満たしている場合には、コストパフォーマンスを重視したプランのご用意があります。一部作業を提携ラボで行い、定型のサービスでコストを抑えます。
低価格RNA-Seqはこちら
RNA-Seq(RNAシーケンス)とは
- RNA-Seq(RNAシーケンス)は、次世代シーケンサーを用いてメッセンジャーRNA(mRNA)やlincRNA、non-coding RNAなどの配列情報を網羅的に読み取ります。その得られた配列情報から、どのRNAが発現しているか数値(発現値)に変換することで遺伝子発現を調べる解析手法です。
- ストランド特異的に解析するので、ゲノムDNAの2本鎖のどちらからmRNAが生成されたのかを知ることが出来ます。すなわち、センス鎖、アンチセンス鎖を区別した解析が可能です。
- マイクロアレイでは解析できない、エクソンレベルでの発現差解析、融合遺伝子の検出、mRNAからの変異解析など豊富な情報が得られます。
- 細胞や組織由来のRNAのほか、分解が進んだFFPE(ホルマリン固定パラフィン包埋)由来RNA、血清・血漿由来RNA、エクソソーム由来RNA、ソーティング細胞由来RNA、RIP-Assay後のRNAなど、様々なサンプルに対応しています。
解析の目的に応じたライブラリー調整
(PolyA mRNA 精製)
Oligo dTビーズを用いて、PolyAの付いたRNAを精製し、ランダムプライマーでcDNA合成してシーケンスライブラリを調整するので、mRNAに特化した解析が可能です。
ただし、分解が進んでいないRNAであることが重要になります。分解が進んでいると、PolyAの付いたRNAも断片化されてしまうので、PolyAの付いたRNAを精製するのが困難になるためです。
RNAの分解が進んでいる場合は、rRNA除去をする方法をおすすめします。
また、cfRNA(セルフリーRNA)の解析がメインの場合(血漿・血清・培養上清・エクソソームなどから抽出したRNAで解析する場合)も、rRNA除去をする方法をおすすめします。
(rRNA除去)
対応生物種は、ヒト、マウス、ラットまたはバクテリアです。
total RNAにはrRNA、tRNA、mRNA、non-cording RNA、cfRNAなど様々なRNAが含まれますが、細胞由来のtotal RNAに最も大量に含まれるrRNA(リボソームRNA。約8割含まれるといわれる)を除去し、rRNAを除いたRNAを解析する方法です。
rRNAの除去には、rRNAの配列に相補的な配列を持つプローブを用いて除去します。
ヒト、マウス、ラットまたはバクテリアに対応したrRNA除去試薬を使用するので、その他の生物種では、除去効率が低くなります。
また、血漿・血清・培養上清・エクソソームなどから抽出したRNAを用いて、cfRNA(セルフリーRNA)の解析を目的とする場合も、この方法をおすすめします。
対応生物種は、ヒト、マウス、ラットです。rRNAを除いたRNAを解析する方法です。
rRNAの除去には、rRNAの配列に相補的な配列を持つプローブを用いて除去しますが、分解が進んだFFPE由来のRNAでも実施可能です。
ただし、DV200が30%以下のサンプルでは解析が困難です。
FFPEの作製には、中性緩衝性ホルマリンを使用すること、ブロックから薄切後、時間を空けずにRNA抽出することをおすすめします。
rRNAを除いたRNAを解析する方法で実施します。
分解が進んだRNAでも実施可能ですが、高度に分解が進んでいる場合は解析が困難になります。
1~500cellsの場合、RNA抽出をせずに、細胞を溶解してダイレクトにライブラリ調整をする方法もありますが、酵素反応を阻害するバッファーの混入があると、Genome DNAを除去しきれず、RNAの解析が困難になります。
血液試料をグロビンmRNAを除去をせずにRNA-Seqを実施した場合、血液中に大量に含まれる赤血球由来のグロビンmRNAが含まれるため、シーケンスデータの約1/4程度がグロビン遺伝子ファミリーに割り当てられる傾向があります。このため、グロビンmRNAを除去することをおすすめします。
サービス概要

サンプルのクオリティチェック


シーケンスライブラリー調製


シーケンス


データ解析
- サンプルのクオリティチェック(QC)を行います。
-
サンプルの種類、分解度合い、解析の目的に応じたライブラリー調製キットを使用し、
total RNAまたはmRNAからcDNA合成後、シーケンスアダプターを付加したシーケンスライブラリーを調製します。
ライブラリー調製キット(Library Kit)は下記のものを取り扱っております。
ご希望のキットがありましたらご相談ください。
前処理試薬
- QIAseq FastSelect –rRNA HMR Kit(QIAGEN社)
-
QIAseq FastSelect –Globin Kit(QIAGEN社)
-
QIAseq FastSelect –rRNA/Globin Kit(QIAGEN社)
-
NEBNext Poly(A) mRNA Magnetic Isolation Module(NEB社)
-
NEBNext rRNA Depletion Kit(NEB社)
-
TruSeq Stranded total RNA Ribo-Zero H/M/R(illumina社)
-
TruSeq Stranded total RNA Ribo-Zero H/M/R Gold(illumina社)
- TruSeq Stranded total RNA Ribo-Zero Globin(illumina社)
- NEBNext Ultra II Directional RNA Library prep for Illumina(NEB社)
-
SMART-Seq Stranded Kit(Clontech社)
- TruSeq stranded mRNA(illumina社)
- 調製したシーケンスライブラリーはQC後、illumina社の次世代シーケンサー(NextSeq 500, NovaSeq 6000, Hiseq Xなど)でシーケンスします。
-
シーケンス後のデータは、リファレンスゲノムにアライメント(マッピング)し、発現量を定量化(正規化)し、アノテーション情報を含むExcel形式のファイルで納品します。なお、デフォルトでは、TPMで定量化します。その後、 Fold Change (およびt検定) による発現変動遺伝子抽出、階層的クラスタリング 、主成分分析 (PCA)などを行います。
ご希望に応じて、GO解析、パスウェイ(Pathway)解析、融合遺伝子の検出、スプライシングバリアントの検出、転写産物単位での発現差解析などの解析を実施します。
-
GO解析
抽出した遺伝子リストについてGO解析で特徴的なGO Termを抽出します。複数ある遺伝子の特徴を客観的につかむことが出来ます。 -
Pathway解析
Wikipathwaysに登録されたPathway上に、解析で得た遺伝子を発現情報(UP/DOWN)で色分けしPathway画像を作成します。 さらに変動した遺伝子を有意に多く含むPathwayを絞り込むことができます。
-
GO解析
データ解析

-
1.リード品質の評価
-
2.リファレンスゲノムへの
アライメント(マッピング)-
3.発現データの正規化
-
4.階層的クラスタリング
&主成分分析(PCA)-
5.発現変動遺伝子抽出
-
6.GO解析
- 7.Pathway解析
-
6.GO解析
-
5.発現変動遺伝子抽出
-
4.階層的クラスタリング
-
8.融合遺伝子の検出
-
9.新規スプライス部位の検出
-
10.エクソンレベル、
スプライシングレベルの
発現差解析
-
10.エクソンレベル、
-
9.新規スプライス部位の検出
-
3.発現データの正規化
-
2.リファレンスゲノムへの
- リードの品質の評価を行います。生のリードデータから、アダプター配列やクオリティの悪い塩基をトリミング (除去)を行います。
- リファレンスゲノムへのアライメント(マッピング)を行います。全リード数の集計、およびアライメント(マッピング)した全リード数、染色体別リード数を集計します。
- サンプル間のリードカウントを補正するために正規化(TMM、TPM等)を行います。
- 階層的クラスタリングと主成分分析(PCA)を行います(6解析以上の場合)。
- ご指定の比較に沿って、変動する遺伝子を抽出します。
- 発現変動遺伝子についてGO解析で特徴的なGO Termを抽出します。
- 得られた全遺伝子をPathway上にマッピングし、Pathway画像を作成します。発現変動遺伝子を有意に多く含むPathwayを絞り込むことが出来ます。
- 融合遺伝子の検出を行います。
- 新規スプライス部位の検出を行います。
- エクソンレベル、スプライシングレベルの発現差解析などを行います。
納品物

- original_fastq (FASTQ生データ)
- trimmed_fastq (トリミング済みFASTQ)
- bam (アライメントデータ)
- サンプルQC結果
- データQC結果(FastQC、MultiQC)
- データ解析結果(正規化、変動遺伝子抽出、GO解析、Pathway解析)
※USBメモリ等の記憶媒体もしくはクラウド経由で納品します。
-
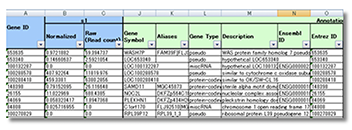
正規化データ(エクセル)
-
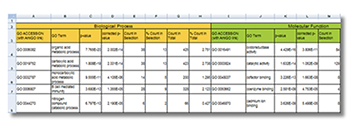
GO解析結果(エクセル)
-
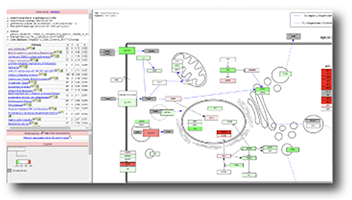
Pathway解析結果
-

ストランドを考慮した発現解析
納期

-
サンプルQC通過後、2か月程度。
(データ解析を含む場合は2.5か月程度)
サンプルについて(RNA-Seqサービス)

| RNA量 | OD260/280 | RIN値 | 送付液量 | DV200 | |
|---|---|---|---|---|---|
| total RNA | 100ng | 1.5 以上 | 7 以上 | 10μL 以上 |
- |
| FFPE 由来 total RNA |
500ng以上 | - | - | 10μL 以上 |
50以上 |
| 微量 total RNA |
10pg ~10ng | - | - | 10μL 以上 |
- |
| 細胞数 | 備考 | ||||
| 微量細胞 | 1 ~ 500 cells | 事前にサンプリングの方法についてお打ち合わせが必要です。必ず予めご相談ください。 | |||
ポイント
- 有機溶媒が混入していない、カラム精製をしたtotal RNAをご送付ください。
- RNAは DNase 処理を行い、DNA のコンタミネーションがない状態のサンプルをご送付ください。
- total RNAはNuclease Free Waterに溶解してください。
注意
- 国内ラボで実施の場合。提携企業先にサンプルを送付する場合はサンプル要件が異なり、より多くのRNA量を必要とします。低価格RNA-Seqサービスをご参照ください。
- サンプル量が上記に満たない場合はお気軽にご相談ください。
- 組織、細胞、FFPEスライド等は、DNA/RNA抽出をご確認いただくか別途お問い合わせください。
- FFPE由来ではない分解サンプルは解析が難しい場合があります。
- 送付方法はサンプル送付方法をご確認ください。
低価格RNA-Seqサービス

特徴(低価格RNA-Seqサービス)
- コストパフォーマンスを重視した実験解析プランのご用意もあります。定型のサービスでコストを抑えます。
- 分解のないtotal RNA 1μgから受け入れます。
一部作業を提携企業で行います。このためレポートの一部が英語となります。
実験内容に関するご質問についてお答えに時間がかかることがあります。
サンプル要件を十分に満たしていない場合はデータの保証をいたしかねます。
サンプルについて(低価格RNA-Seqサービス)
| RNA量 | RIN値 | rRNA ratio | |
|---|---|---|---|
| total RNA | 1μg | 7 以上 | 1.0以上 |
ポイント
- 有機溶媒が混入していない、カラム精製をしたtotal RNAをご送付ください。
- RNAは DNase 処理を行い、DNA のコンタミネーションがない状態のサンプルをご送付ください。
- total RNAはNuclease Free Waterに溶解してください。
注意
Contact
お問い合わせ
受託サービス・取扱製品に関するお見積もり、ご依頼、お問い合わせは下記より担当窓口へお問い合わせください。

