NOIR-SS
NOIR-SSとは

NOIR-SS (Non-overlapping Integrated Read Sequencing System) は分子バーコード技術を利用した次世代シーケンスによるがん関連遺伝子パネル解析システムです。
特徴
-
分子数の絶対定量
従来法では無視されている分子バーコード上のエラーを除去し、高精度に希少変異を検出。 -
高感度
高精度アッセイと独自のノイズ除去効果により、0.1%の希少変異まで検出が可能。 -
癌腫ごとに最適化されたパネル
最適化パネル : 肺がん及び膵臓がん
例として、肺がんパネルでは、EGFR-TKIの治療効果に関わるコンパウンド変異を高感度かつ網羅的に検出可能。 -
対象検体
血中遊離DNA、凍結検体、FFPE、胸水や細胞浮遊液
検体種別に対応したQC、アッセイ系の最適化を実施しています。 -
カスタムパネル構築が可能
とらえたい変異を高感度に検出するために、パネル及びアッセイの最適化を行います。
実績のあるカスタムパネルの例 : EGFRパネル、TP53パネル
背景
近年、非侵襲的に腫瘍細胞由来の遺伝子変異をモニタリングするリキッドバイオプシーへの需要が高まってきています。次世代シーケンス技術は血中遊離DNAの遺伝子変異を網羅的に検出できますが、その際シーケンスエラーやPCRに伴うエラーが問題となります。これらのエラーと腫瘍由来の希少変異を区別する方法として、分子バーコード技術が注目されています。NOIR-SSは独自の分子バーコード技術により、正確な遺伝子変異情報を提供します。
用途
- がんコンパニオン診断薬開発
- がん遺伝子パネルによるクリニカルシーケンス
- リキッドバイオプシー応用
※大阪府立成人病センターとDNAチップ研究所との共同研究の成果に基づいています。原著論文等はこちらをご参考ください。
技術背景

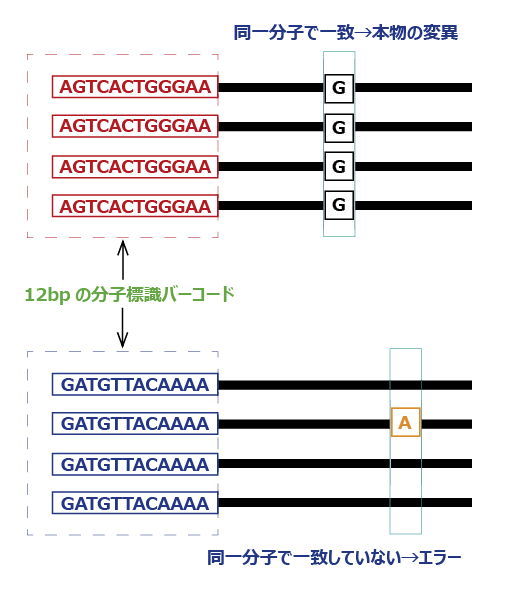
分子バーコードによる変異解析
PCR前に各DNA分子にバーコードを付加して、次世代シーケンスにより配列を大量に読み取ります。同一分子バーコードの配列コンセンサスを見て、シーケンス/PCRエラーとレア変異を区別することができます。
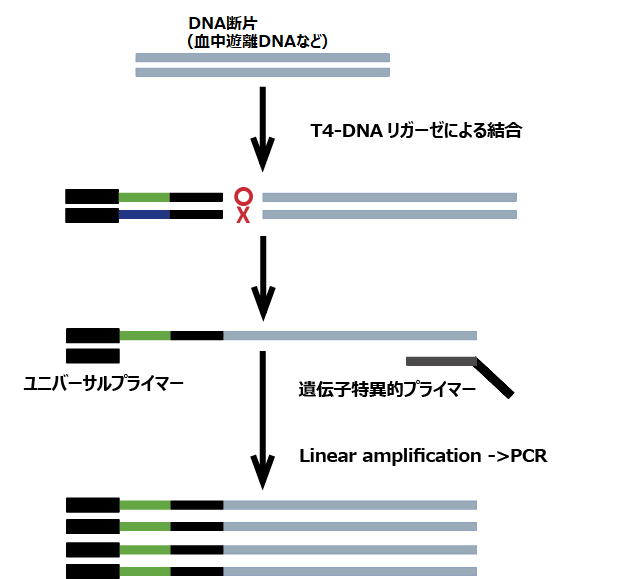
NOIR-SSにおける遺伝子増幅
DNA断片に分子バーコード付きのアダプターを付加して、遺伝子特異的プライマーとユニバーサルプライマーでPCR増幅します。片側のみ特異的プライマーのため、バイアスがより少なく、融合遺伝子検出にも強いシステムを採用しています。
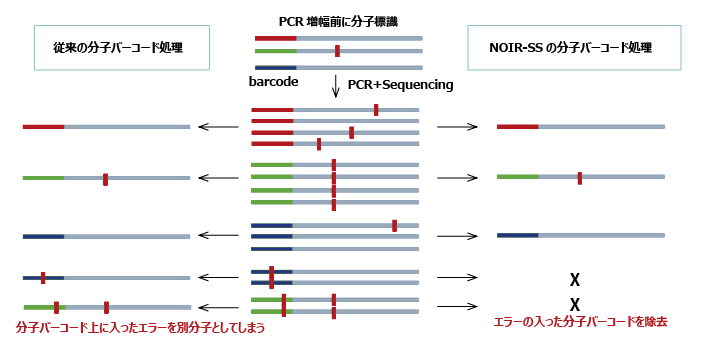
特徴

独自の分子バーコード技術
NOIR-SSの分子バーコード技術の特徴
PCR前に分子の標識となるバーコードを付加して、PCR増幅後、次世代シーケンスにより読み取ります。同一分子バーコードを有するDNA配列のコンセンサスをみることで、シーケンス/PCRエラーとレア変異を区別することができます。NOIR-SSでは、従来の方法では無視されている分子バーコード上のエラーを除去することで分子数を絶対定量し、高精度に希少変異を検出します。
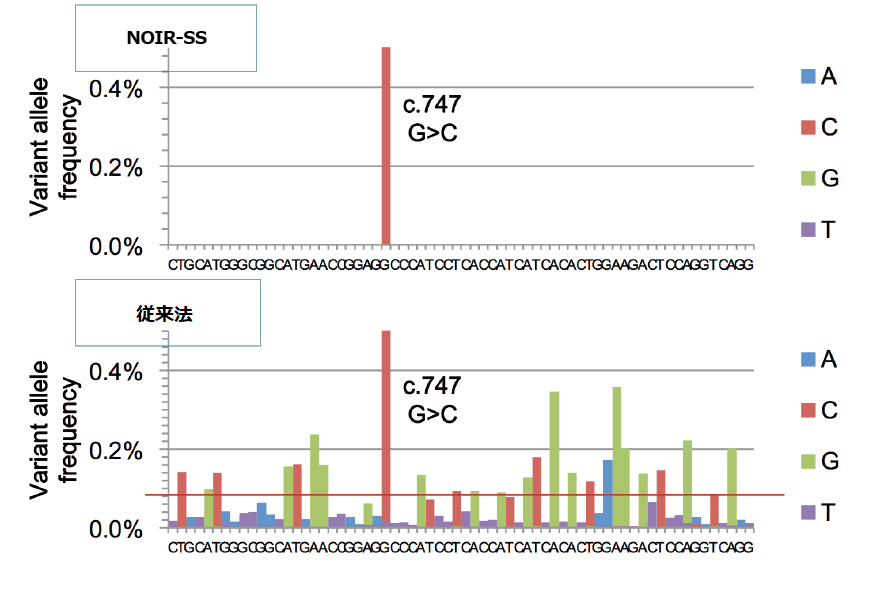
TP53遺伝子上(横軸は塩基サイト)における、
変異率
分子バーコード処理を行わない場合、c747G>C変異以外に多くの偽陽性が検出されてしまいますが、NOIR-SSによる分子バーコード処理により、バックグラウンドノイズは取り除かれ、シグナル/ノイズ比が大幅に改善します。
※NOIR-SSの分子バーコード解析技術は、大阪府立病院機構とDNAチップ研究所による共同研究成果として特許を取得しております
近年、非侵襲的に腫瘍細胞由来の遺伝子変異をモニタリングするリキッドバイオプシーへの需要が高まってきています。次世代シーケンス技術は血中遊離DNAの遺伝子変異を網羅的に検出できますが、その際シーケンスエラーやPCRに伴うエラーが問題となります。これらのエラーと腫瘍由来の希少変異を区別する方法として、分子バーコード技術が注目されています。NOIR-SSは独自の分子バーコード技術により、正確な遺伝子変異情報を提供します。
設計済み遺伝子パネル

遺伝子パネルの種類
2種類のパネルがあります。下記の遺伝子に限らずカスタムで設計することも可能です。
| 肺癌パネル | 膵臓癌パネル |
|---|---|
| ALK | CDKN2A |
| BRAF | CTNNB1 |
| EGFR | GNAS |
| ERBB2 | HRAS |
| KRAS | KRAS |
| MET | NRAS |
| TP53 | SMAD4 |
| NFE2L2 | TP53 |
| PIK3CA |
上記掲載遺伝子にかぎらず、カスタムな遺伝子パネル設計が可能です。
論文

High-fidelity target sequencing of individual molecules identified using barcode sequences: de novo detection and absolute quantitation of mutations in plasma cell-free DNA from cancer patients.
DNA Res. 2015 Aug;22(4):269-77. doi: 10.1093/dnares/dsv010. Epub 2015 Jun 29.
Yoji Kukita1, Ryo Matoba2, Junji Uchida3, Takuya Hamakawa1,4, Yuichiro Doki4, Fumio Imamura3, and Kikuya Kato1
- Department of Molecular and Medical Genetics, Research Institute, Osaka Medical Center for Cancer and Cardiovascular Diseases
- DNA Chip Research Inc.
- Department of Thoracic Oncology, Osaka Medical Center for Cancer and Cardiovascular Diseases
- Department of Gastroenterological Surgery, Osaka University Graduate School of Medicine
https://academic.oup.com/dnaresearch/article/22/4/269/348206
Selective identification of somatic mutations in pancreatic cancer cells through a combination of next-generation sequencing of plasma DNA using molecular barcodes and a bioinformatic variant filter.
PLoS One. 2018 Feb 16;13(2):e0192611.doi: 10.1371/journal.pone.0192611. eCollection 2018.
Yoji Kukita1, Kazuyoshi Ohkawa2, Ryoji Takada2, Hiroyuki Uehara2, Kazuhiro Katayama2, Kikuya Kato1
- Department of Molecular and Medical Genetics, Research Institute, Osaka Medical Center for Cancer and Cardiovascular Diseases
- Department of Hepatobiliary and Pancreatic Oncology, Osaka Medical Center for Cancer and Cardiovascular Diseases
https://journals.plos.org/plosone/article?id=10.1371/journal.pone.0192611
Contact
お問い合わせ
検査に関するご依頼、お問い合わせは下記より担当窓口へお問い合わせください。

